|
|||
|
| ARG |
Diagnóstico pre y postnatal de enfermedades genéticas
1- Weerakkody RA, Vandrovcova J, Kanonidou C, Mueller M, Gampawar P, Ibrahim Y, et al. Targeted next-generation sequencing makes new molecular diagnoses and expands genotype-phenotype relationship in Ehlers-Danlos syndrome. Genet Med. 2016;18(11):1119-1127. 2- Bhoj EJ, Yu Z, Guan Q, Ahrens-Nicklas R, Cao K, Luo M, et al. Phenotypic predictors and final diagnoses in patients referred for RASopathy testing by targeted next-generation sequencing. Genet Med. 2016 Oct 20. doi: 10.1038/gim.2016.169. 3- Bastida Bermeja JM, González-Porras JR, Jiménez C, Benito R, Ordoñez GR, Álvarez-Román MT, et al. Application of a molecular diagnostic algorithm for haemophilia A and B using next-generation sequencing of entire F8, F9 and VWF genes. Thromb Haemost. 2016 Oct 13;117(1). 4- Yoshida S, Kido J, Matsumoto S, Momosaki K, Mitsubuchi H, Shimazu T, et al. Prenatal diagnosis of Gaucher disease using next-generation sequencing. Pediatr Int. 2016 ;58(9):946-9. 5- Faria R, Silva B, Silva C, Loureiro P, Queiroz A, Fraga S, et al . Next-generation sequencing of hereditary hemochromatosis-related genes: Novel likely pathogenic variants found in the Portuguese population. Blood Cells Mol Dis. 2016;61:10-5. 6- Cecconi M, Parodi MI, Formisano F, Spirito P, Autore C, Musumeci MB, et al. Targeted next-generation sequencing helps to decipher the genetic and phenotypic heterogeneity of hypertrophic cardiomyopathy. Int J Mol Med. 2016;38(4):1111-24. En español 1- Martínez Barrios E, Martínez Puchol AI, Roset A, Zambón D, Clària J, Bernat M. Aplicación de la secuenciación masiva de nueva generación al diagnóstico molecular de la hipercolesterolemia familiar. Revista del laboratorio clínico. 2015;8(1):8-18. 2- Jiménez-Escrig A, Gobernado I, Sánchez-Herranz A. Secuenciación de genoma completo: un salto cualitativo en los estudios genéticos. Rev Neurol 2012; 54 (11): 692-8. 3- Palacios-Verdú MG, Pérez-Jurado LA. Nuevas metodologías en el estudio de enfermedades genéticas y sus indicaciones. Pediatr Integral 2014;18 (8): 515-28. Agudice su ingenio
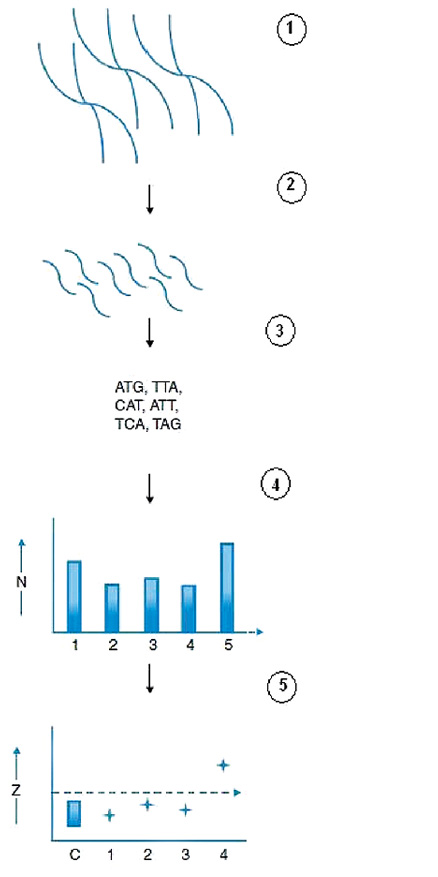
Detección de aneuploidías fetales empleando secuenciación de nueva generación. En este procedimiento inicialmente se aíslan los fragmentos de ADN fetal libres en el plasma materno y se produce una librería con secuencias adaptadoras especiales. Estos adaptadores permiten el consiguiente análisis múltiple. Luego la librería se somete a secuenciación masiva para determinar la secuencia de cada fragmento. Las secuencias se alinean con el genoma de referencia y se identifica su localización cromosómica mediante métodos bioinformáticos. A continuación se cuenta el número de reads para cada cromosoma. Para el cromosoma 21 normalmente se obtienen varios miles de reads, los cuales pueden entonces compararse con los otros millones de reads distribuidos a lo largo del genoma. Si el feto está afectado con síndrome de Down el número de reads asignados al cromosoma 21 será ligeramente mayor en comparación con el número obtenido a partir de fetos normales. La comparación de estos datos con un banco de muestras de referencia y el empleo de valores prefijados de corte (Z score) permite determinar la dotación cromosómica. (Adaptado de Hanh et al 2011) A- Extracción ADN y preparación librería Le informamos que se le obsequiará un CD con material bibliográfico a elección de un listado que tenemos disponible, a los primeros que nos hagan llegar su respuesta correcta al correo electrónico bibliote@fbpba.org.ar. web: http://www.faba.org.ar |
||
| Subir | ^ |
Diagnóstico pre y postnatal |
Novedades: Definiendo términos |