|
|||
|
| ARG |
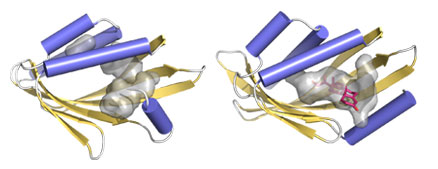
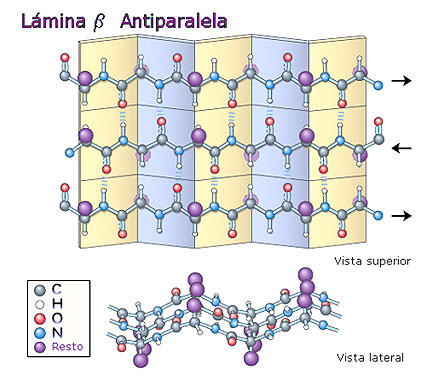
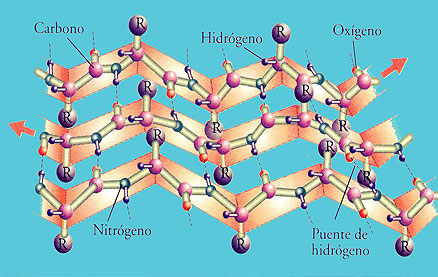
Describen los principios para diseñar proteínas de utilidad clínica Por Ana M. Pertierra Considerando que en las proteínas naturales los sitios activos y las cavidades donde se unen los ligandos están formados a menudo por estructuras de lámina beta curvas, los investigadores de Seattle se plantearon el desafío de dilucidar los mecanismos para el diseño de proteínas hechas a medida para unirse a ligandos específicos.
FABAINFORMA se comunicó a través del correo electrónico con Benjamín Basanta, un joven biólogo argentino que participó de este estudio (http://science.sciencemag.org/content/355/6321/201), y que se encuentra dedicado a su tesis doctoral en este laboratorio universitario norteamericano liderado por un especialista en diseño proteico y profesor de Bioquímica, el Dr. David Baker. • En el trabajo que el grupo de investigación del Departamento de Bioquímica de la Universidad de Washington acaba de publicar en Science, han logrado las reglas o principios para el diseño de ciertas proteínas con una conformación espacial particular, con cavidades o bolsillos, a partir de láminas beta ¿Cuál es la importancia de este logro y cuál su implicancia práctica para futuras investigaciones? Los principios que describimos en este trabajo nos permitieron crear, desde cero, proteínas con una gran superficie cóncava que da lugar a cavidades y bolsillos en su interior. Antes de que conociéramos dichos principios, era imposible diseñar estructuras con láminas beta curvas, lo cual limitaba el repertorio de superficies cóncavas y bolsillos a unas pocas variedades de proteínas diseñadas desde cero o las que ya existen en la naturaleza. Esperamos que estos principios allanen el camino para la creación de proteínas novedosas, en las cuales las cavidades y bolsillos son diseñados para retener o detectar moléculas de interés como hormonas, fármacos o sustancias tóxicas. • ¿Cuál ha sido el máximo obstáculo para la obtención de proteínas de novo con estructuras similares a las de la naturaleza? El problema de cómo reproducir muchas de las características presentes en proteínas naturales es que no comprendemos cuales son los factores que las estabilizan o, aun conociendo estos factores, no se han desarrollado algoritmos para diseñarlas. En el caso de nuestro trabajo, tuvimos que resolver ambos problemas, aunque implementar un algoritmo de diseño fue relativamente fácil una vez que establecimos los principios.
•¿Qué propiedades tienen las proteínas naturales con "cavidades" o plegamientos, cuáles sus funciones biológicas y de qué forma se podría aplicar la tecnología desarrollada por ustedes para hacer aportes a la biología y a la salud humana? En muchas estructuras proteicas las cavidades y bolsillos suelen funcionar como sitios activos, donde se aloja una molécula y/u ocurre una reacción. Parte de la ventaja evolutiva de tener estos bolsillos, aislados del solvente circundante, es que permiten reconocer con gran especificidad sustratos o moléculas señal, como las hormonas, y, si el bolsillo es un sitio capaz de catalizar reacciones, conferir gran especificidad a la estereoquímica de dicha reacción. Los principios que describimos abren el camino para crear proteínas con una gran variedad de bolsillos, en los que pueden diseñarse las interacciones específicas para cada caso. El reconocimiento ultraespecífico de moléculas señalizadoras en sistemas biológicos podría ser muy útil en el diagnóstico de enfermedades humanas y animales. Muchas veces, la estereoquímica de los productos intermedios en la síntesis de fármacos u otras moléculas útiles es difícil de controlar y puede resultar en menores márgenes de ganancia y gran cantidad de desechos. El diseño a medida de catalizadores proteicos podría usarse para solucionar estos problemas. • ¿Con qué metodología han trabajado para arribar a estos resultados? La etapa inicial de análisis de proteínas naturales se hace completamente en computadora, mediante programación, estadística básica y observación de modelos atómicos 3D de algunas estructuras. La etapa de diseño también se hace en computadora, utilizando el paquete de software Rosetta Molecular Modeling, desarrollado inicialmente en este laboratorio y luego por una comunidad de grupos de investigación alrededor del mundo. Los cálculos hechos con este software pueden hacerse en una computadora de escritorio, pero para aumentar el número y velocidad de muestreo utilizamos clusters de cómputo de nuestro laboratorio, de la Universidad de Washington y hasta tiempo de cómputo donado por personas de todo el mundo a través del proyecto Rosetta@home. La etapa final es la de producción de los diseños, que se hace mediante técnicas de ADN recombinante en bacterias. Las bacterias son genéticamente modificadas para producir la proteína, luego se las licúa para liberar el contenido y purificar la proteína de interés. Los ensayos bioquímicos de estabilidad y estructura cuaternaria se hacen con la proteína purificada. Parte de la muestra de proteína es luego disuelta en un buffer en el que forma cristales, de los cuales se puede obtener la estructura atómica para confirmar que efectivamente el diseño es recapitulado en la realidad. • Egresado como biólogo de la FCEN de la UBA, usted está trabajando en este tema de diseño de proteínas en el laboratorio de la Universidad de Washington para su tesis doctoral. ¿Desde cuándo está allí y hasta cuándo estima que estará? Entré a la Universidad de Washington como estudiante de doctorado en septiembre de 2013, y trabajo en el laboratorio de David Baker desde junio de 2014. En promedio, el tiempo para una tesis doctoral en Estados Unidos es de cinco o cinco años y medio, con lo cual me quedarían dos años más para seguir investigando en el tema y tratar de llevar este proyecto al objetivo final, que es utilizar estos principios para crear proteínas con funciones como las que mencioné anteriormente.
|
||
| Subir | ^ |
 |
Un paso hacia el cultivo en cerdos de órganos humanos |
 |
Describen los principios para diseñar proteínas de utilidad clínica |