El veredicto de la genética
La identificación genética de especies y su uso en el control de alimentos, conservación de la biodiversidad y detección de fraudes.
El Comité de Redacción de Acta Bioquímica Clínica Latinoamericana ha seleccionado este artículo publicado en la Revista CIENCIA HOY, Volumen 17 Nº 98 – 2007, para su difusión a través de FABA Informa.
Diego M. Posik, María Verónica Ripoli, Pilar Peral García y Guillermo Giovambattista
Centro de Investigaciones en Genética Básica y Aplicada (CIGEBA),
Facultad de Ciencias Veterinarias, UNLP.
En marzo de 1996 se produjo en Gran Bretaña un brote del mal de la vaca loca –o encefalopatía espongiforme bovina (EEB)– por el que resultaron infectados varios miles de animales. Ello originó una alarma generalizada en la población, ya que dicha enfermedad, producida por una proteína infecciosa llamada prión, que ataca el cerebro de los vacunos, tiene en el hombre su equivalente fatal en la enfermedad de Creutzfeldt-Jacob.
Los bovinos británicos se infectaron porque fueron alimentados con harina de hueso y suplementos proteicos que contenían cerebros molidos de oveja. En las ovejas y cabras tiene carácter endémico en ese país otra encefalopatía espongiforme, llamada scrapie, de la que no había evidencia de contagio a las personas y que, se creía, tampoco se transmitía a otras especies animales. Sin embargo, los casos de mal de la vaca loca de 1996 llevaron a la convicción de que se había producido el pasaje de la enfermedad de ovinos a vacunos, o de una especie animal a otra, y esto preocupó a los investigadores, sobre todo porque, por los mismos años, había aumentado el número de tamberos que murieron a causa de la enfermedad de Creutzfeldt-Jacob. La conclusión fue que los priones infecciosos podrían pasar de la hacienda vacuna a los seres humanos, algo que con el tiempo se confirmó.
Una vez conocidos los contagios de la enfermedad neurodegenerativa de ovinos a vacunos, es decir, a través de las barreras que separan las especies, las legislaciones de la Unión Europea y de los Estados Unidos prohibieron el uso de proteínas derivadas de tejidos de rumiantes para alimentar animales de consumo humano. Además, se intensificaron las inspecciones tendientes a controlar los componentes utilizados en la elaboración de alimentos balanceados para rumiantes, con el fin de detectar la presencia de materias primas procedentes de animales prohibidos (entre los que se encuentran bovinos, ovejas, cabras, ciervos y alces), de suerte de prevenir la propagación de la EEB.
PCR, la reacción en cadena de la polimerasa
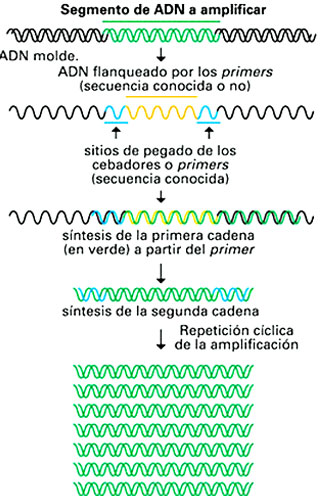
< La reacción en cadena de la polimerasa (o PCR, sigla de Polymerase Chain Reaction) es una técnica ideada por Kary Mullis en 1986. Se utiliza para sintetizar in vitro secuencias específicas de ADN. Las polimerasas son enzimas que sintetizan a los ácidos nucleicos, (tanto el ADN y el ARN). El método imita el proceso natural de replicación del ADN en las células, llevado a cabo por polimerasas de ADN. El mayor mérito de la PCR es que, a partir de un pequeño número de moléculas, permite obtener gran número de copias de un fragmento de ADN, lo que resulta de enorme utilidad en gran parte de los estudios de biología molecular. El tamaño del fragmento replicado puede variar entre unos 100 y unos pocos miles de pares de bases.
Para realizar la multiplicación de un fragmento determinado de ADN se debe contar con una cadena que funcione como molde a amplificar y con un par de cebadores o primers. Los últimos son oligonucleótidos (cortos tramos de ADN de cadena simple, compuestos por aproximadamente unas 20 bases) de secuencia conocida que se unen a los extremos del fragmento a amplificar. Si bien es necesario conocer la secuencia de bases de los cebadores, no es preciso conocer la del interior del segmento a amplificar.
La PCR puede utilizarse para la detección de mutaciones (por ejemplo, las que producen enfermedades hereditarias), para identificar secuencias específicas propias de infecciones por virus o bacterias, para reconocer especies, para análisis de filiación y para estudiar ADN fósil, entre otras aplicaciones.
En paralelo con lo anterior –y posiblemente en parte por su influencia–, en los últimos años han cambiado muchas actitudes de los consumidores, principalmente en los países desarrollados: buscan la comida que consideran más sana y procuran conocer el origen y la calidad de los productos que compran. No sólo se proponen evitar alimentos adulterados, sino que se ha creado en muchos lugares una resistencia a los elaborados con organismos transgénicos.
Cabe preguntarse, ante esta situación, si la genética puede ayudar a garantizar la seguridad alimenticia de la población. ¿Es posible extraer ADN de alimentos procesados? Y, si así fuera, ¿se pueden identificar los componentes de un alimento balanceado, para evitar aquellos asociados con la diseminación de la EEB?
Las técnicas genéticas han demostrado ser de gran utilidad para resolver, entre otras, dos tipos de situaciones: la identificación de los individuos de los que provienen las muestras que se analizan, y la determinación de la especie a que esos individuos pertenecen. Los primeros análisis utilizados para identificar especies, anteriores a la moderna genética molecular, se valieron de métodos microscópicos, orientados al estudio de fragmentos de huesos de animales. Tal tipo de análisis consume mucho tiempo, requiere la presencia de especialistas y sólo permite determinar la clase zoológica (mamífero, ave, pez) pero no la especie de la que provino una muestra.
La aparición de métodos genéticos brindó una mejor alternativa, pues estos permiten llegar a conclusiones con alto grado de especificidad. Además, son aplicables incluso en el caso de productos sometidos a altas temperaturas durante su elaboración, como ocurre con los alimentos balanceados. Se ha demostrado que, si bien las proteínas se degradan cuando se cocinan los alimentos, el ADN queda suficientemente inalterado, pues es una molécula relativamente resistente a la temperatura y puede ser identificada aún después del proceso térmico.
Entre los primeros casos en los que se aplicaron métodos basados en el ADN para resolver esta clase de problemas pueden mencionarse el control de stocks pesqueros, la detección de tráfico de especies silvestres en peligro de extinción y la identificación del origen geográfico del atún enlatado, lo último para resolver una controversia entre pescadores del Pacífico y del Atlántico relacionada con el uso de redes que atrapan a delfines.
¿En qué se basan los métodos de identificación de especies mediante el análisis del ADN? Las distintas especies de vertebrados tienen, en esencia, los mismos genes. En otras palabras, sus respectivos genomas (el conjunto de todos sus genes) coinciden, lo cual se debe a que comparten una historia evolutiva y descienden de un ancestro común. Como las proteínas sintetizadas a partir de genes homólogos (genes con cierta semejanza debido a que han sido heredados de un ancestro común) cumplen las mismas funciones en las diferentes especies, tienden a mantener su estructura y función a pesar de la acumulación de mutaciones producida desde la separación de la descendencia del ancestro común en especies diferentes. Por esta razón, cuando se comparan las secuencias de genes homólogos correspondientes a diferentes especies es posible advertir regiones conservadas de las moléculas de ADN de su genoma (en las que no se advierten diferencias entre las secuencias de nucleótidos de las diferentes especies) y regiones variables (en las que se advierten esas diferencias entre las especies) (figura 1).
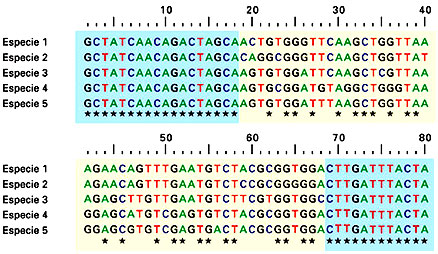
Figura 1.
Comparación de secuencias de una misma región de ADN de 5 especies diferentes. Las regiones conservadas y variables están sombreadas en colores celeste y amarillo respectivamente. Los asteriscos identifican las columnas en las que no hay diferencia entre las especies.
¿Cuáles son las etapas de aplicación de esta técnica? El primer paso para la detección del origen específico de los componentes de un alimento consiste en la extracción y purificación de moléculas de ADN. Cuando los alimentos son frescos y han sido mantenidos a bajas temperaturas, el material genético permanece en muy buenas condiciones. Por el contrario, cuando se analizan productos procesados, como los alimentos balanceados, los embutidos y los quesos, podría esperarse que el ADN esté degradado y que no sea posible su identificación. Sin embargo, aunque esto es hasta cierto punto cierto, hoy existen técnicas que permiten purificar y caracterizar moléculas de ADN con algún grado de degradación.
El segundo paso consiste en amplificar pequeños fragmentos seleccionados del ADN por medio de la reacción en cadena de la polimerasa, o PCR. El análisis se puede realizar por dos caminos diferentes. Uno se vale de cebadores (o primers) universales, que permiten amplificar fragmentos de ADN de cualquier especie de mamíferos. Posteriormente, se realiza la digestión de dichos fragmentos con enzimas de restricción, que cortan el ADN en secuencias específicas (ver recuadro ‘Digestión con enzimas de restrición y separación de moléculas por electroforesis’). El resultado de este proceso consiste en un patrón de bandas, semejante a un código de barras, que es diferente para cada especie debido a las regiones variables (figura 2, panel izquierdo). Los patrones de la digestión obtenidos por esta técnica se comparan con los patrones correspondientes a las especies declaradas por el fabricante del producto analizado y con aquellas que potencialmente podrían estar presentes como contaminantes o adulteraciones.
El otro camino que utiliza la PCR para amplificar pequeños fragmentos seleccionados del ADN recurre a cebadores específicos, o propios de cada especie, diseñados a partir de las regiones variables, lo que resulta en fragmentos de amplificación de tamaños diferentes. La identidad de cada especie se detecta por la presencia o ausencia de bandas (figura 2, panel derecho). Alternativamente, se puede comparar la secuencia de nucleótidos de los fragmentos amplificados con la secuencia conocida de las especies cuya presencia o ausencia se procura constatar (figura 1).
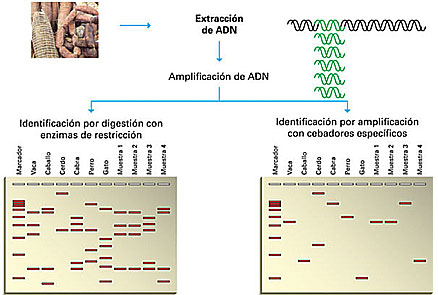
Figura 2.
Identificación genética de los constituyentes de los alimentos. Los primeros pasos implican la extracción de ADN de las muestras y la amplificación de un segmento determinado por la técnica de PCR (en color verde). En la parte inferior del gráfico, a la izquierda, se muestra la separación, por medio de electroforesis, de los fragmentos de ADN resultantes de la digestión con enzimas de restricción de una región del ADN mitocondrial amplificado por PCR. A la derecha se muestra una electroforesis de fragmentos amplificados por PCR utilizando cebadores específicos para cada especie. Los últimos se diferencian por los distintos tamaños de las bandas amplificadas. En ambos paneles se muestran los patrones de bandas característicos de seis especies conocidas comparadas con los patrones de cuatro muestras de origen desconocido.
Para comprender la utilidad práctica de estas técnicas, imagínese el lector que va a un supermercado en busca de queso. En las góndolas encuentra diferentes productos y marcas: las etiquetas anuncian quesos de vaca, de oveja, de cabra, mezclas de estos y hasta una mozzarella de leche de búfala. Si se acerca a la sección de embutidos, verá que algunos proclaman su composición sólo con carne de vaca o de cerdo, otros con ambas carnes, y sospecha que la mortadela contendrá carne equina. No sorprenderá a nadie que los compradores se pregunten si, efectivamente, los productos contendrán los ingredientes declarados en las etiquetas, si habrá adulteraciones y si estarán presentes contaminantes. La identificación de las especies por los métodos explicados de genética molecular constituye una herramienta muy útil para despejar las dudas anteriores, en particular en alimentos como quesos, embutidos, paté, carnes procesadas, harinas de pescado, caviar, conservas, etc. No sólo permite constatar las especies presentes o establecer ausencias, sino, también, determinar las cantidades relativas de los componentes.
El uso de la técnica de PCR como herramienta de cuantificación en el análisis de ADN, inicialmente estuvo restringido al rastreo de organismos genéticamente modificados o a contaminaciones con microorganismos. Es más reciente su uso para evaluar los ingredientes usados en la manufactura de alimentos. Se han creado procedimientos simplificados o estándares para estimar las proporciones de esos ingredientes; por ejemplo, las partes de carne bovina y porcina en un embutido se pueden estimar comparando los resultados de los análisis del producto estudiado con los de muestras control de composición conocida. Recientes modificaciones de la técnica de la PCR (PCR competitivo, PCR en tiempo real) permiten cuantificaciones precisas y confiables de las distintas especies.
La identificación de especies y la certificación de origen tienen importancia para personas que padecen de algunos tipos de alergias, como las producidas, por ejemplo, por proteínas de leche de vaca. Tales pacientes deben sustituir la leche vacuna y sus derivados por productos caprinos u ovinos. Las técnicas que se explicaron permiten distinguir entre especies cercanas, como son vacas, ovejas y cabras, y brindan la posibilidad de detectar procedimientos fraudulentos en la manufactura de esos alimentos, del tipo de la sustitución de una leche por otra. Adulteraciones de esta clase no suelen ser infrecuentes, debido al mayor costo de las leches de cabra y oveja y a sus menores volúmenes de producción. También es posible diferenciar el ADN bovino del de búfalo, algo importante para certificar el origen del queso mozzarella.
Los organismos genéticamente modificados, difundidos en la última década, están cobrando creciente importancia en especies como papa, soja, maíz, bacterias del yogur, etc. Modificaciones genéticas habituales consisten en insertarles genes de otras especies, que pasan a integrar el genoma del organismo en cuestión (el cual, de esa manera, se convierte en un organismo transgénico) y le confieren resistencia a ciertas enfermedades, o al ataque de insectos, o a compuestos químicos como herbicidas. En estos casos, la identificación se realiza mediante la detección de los segmentos de ADN exógeno. El hecho de que ya se comercialicen productos alimenticios que incluyen organismos genéticamente modificados en su composición, y que, por otro lado, existan preocupaciones entre consumidores y entidades ecologistas acerca de su seguridad, han llevado a distintos países a establecer la obligación legal de identificar tales alimentos mediante leyendas apropiadas en sus etiquetas. Los análisis de ADN permiten asegurar la presencia o ausencia de materias primas genéticamente modificadas en los alimentos, tanto a lo largo del proceso de producción como de la comercialización.
También en los alimentos son comunes distintos tipos de contaminaciones bacterianas. Así, en los últimos tiempos ha adquirido resonancia la contaminación de carnes con cepas patógenas de la bacteria Escherichia coli, que producen diarreas severas y causan una grave enfermedad urinaria conocida como síndrome urémico hemolítico. La contaminación de la carne se produce por lo común por falta de higiene, tanto cuando se faenan los animales como durante el procesamiento de los cortes. Es posible utilizar los análisis de ADN para identificar las cepas patógenas y la determinación de en qué etapa del procesamiento y comercialización de la carne se produjo la contaminación.
La caza furtiva y la recolección ilegal de plantas representan una amenaza para una amplia variedad de especies en peligro de extinción, desde grandes felinos, elefantes, osos y rinocerontes, hasta aves, cetáceos, peces y plantas. La mayoría de los países tienen leyes que protegen a los animales y las plantas amenazados. Sin embargo, a veces es difícil obtener evidencia que demuestre la culpa de individuos sospechosos de apropiación o comercio ilegal de especies en peligro de extinción. Los métodos de genética molecular pueden ser usados en tales casos para identificar el origen de material biológico como marfil, cuernos, huevos, caparazones de tortugas, carne, plumas, pelos y materiales vegetales. Estos métodos han adquirido un cometido importante y creciente en la detección y prevención de la caza y el tráfico ilegales de especies silvestres.
Un caso interesante fue la identificación específica de carne de ballenas protegidas en el Japón y en Corea. El análisis de ADN mitocondrial estableció que gran parte de la carne de ballena destinada para consumo humano no era de ballenas Minke, cazadas, según los japoneses, con fines científicos, sino de ballenas de Bryde y de otras dos especies, las azules y las jorobadas. Además, se descubrió que parte de la supuesta carne de ballena no era tal, sino delfín, marsopa, oveja y caballo. No sólo se trataba de un caso de pesca ilegal de ballenas, sino, también, de engaño a los consumidores.
En una situación semejante, el análisis de ADN mitocondrial reveló que el 23% de las muestras de caviar tomadas en Nueva York estaban adulteradas. Este hecho tenía también implicancias conservacionistas, ya que la mayoría de las 27 especies de esturiones se encuentra en peligro de extinción debido a la excesiva pesca y a la degradación del ambiente.
Otros ejemplos de la utilidad de este tipo de técnicas fueron la identificación de antílopes oryx de Arabia (Oryx leucoryx), en peligro de extinción y cazados ilegalmente, y la determinación, por medio del análisis de ADN mitocondrial, de la región donde habían sido cazados 26 chimpancés confiscados en Uganda. Lo último hizo posible que los animales pudieran ser devueltos a su hábitat. Actualmente, se están ideando métodos también basados en ADN mitocondrial para detectar derivados de tigre en medicinas asiáticas.
Las mitocondrias poseen su propio sistema genético, separado y distinto del genoma del núcleo de las células. Según la teoría endosimbiótica (difundida por Lynn Margulis hacia fines de la década de 1960), que se refiere al origen evolutivo de las células eucariotas, este ADN mitocondrial tiene una procedencia diferente del ADN nuclear: provendría de bacterias que desarrollaron una relación simbiótica con células primitivas, precursoras de dichas células eucariotas. El genoma mitocondrial está constituido por moléculas circulares de ADN, como las de las bacterias. Están presentes en varias copias por orgánulo, de forma que en una única célula se pueden encontrar varios miles de copias de estas moléculas. A diferencia del ADN nuclear, el mitocondrial no sufre procesos de recombinación, y posee una tasa de mutación mucho más rápida, que conduce a la acumulación de diferencias, incluso entre especies evolutivamente muy cercanas. Esto, sumado al mencionado alto número de copias, permite que, a partir de unas pocas células o de muestras en las que el ADN ha sufrido alguna degradación, sea posible extraer por lo menos algunas moléculas intactas y así realizar los análisis de identificación de especies.
Digestión con enzimas de restricción y separación
de moléculas por electroforesis
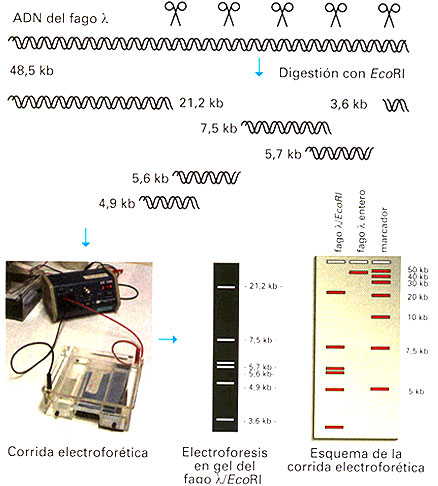
< Una de las primeras herramientas utilizadas en los laboratorios de biología molecular estuvo constituida por las enzimas de restricción (o endonucleasas de restricción), enzimas que cortan el ADN en lugares concretos y específicos de su secuencia. Fueron identificadas en bacterias, en las que aparentemente cumplen una función defensiva frente a la entrada en la célula de un ADN extraño (por ejemplo de un virus).
Debido a que las enzimas, de restricción digieren secuencias específicas de ADN, pueden ser utilizadas para cortar una molécula de este en sitios únicos. Por ejemplo, la enzima EcoRI reconoce la secuencia de seis pares de bases GAATTC, la que está presente cinco veces en el ADN de un virus bacteriano denominado bacteriófago ?. Así, dicha enzima digiere el ADN del virus (o fago) en seis fragmentos de entre 3600 y 21.200 pares de bases de longitud. Los fragmentos pueden separarse según su tamaño por medio de una electroforesis en gel, un método por el que se separan las moléculas basándose en su velocidad de migración en un campo eléctrico. El gel, habitualmente compuesto de agarosa o poliacrilamida, se sitúa entre dos compartimientos con electrodos en los que se dispone un tampón o buffer. La muestra (por ejemplo, una mezcla de fragmentos de ADN para ser analizados) se deposita en ranuras preformadas y se aplica el campo eléctrico. Los ácidos nucleicos tienen carga negativa, por lo que migran al electrodo positivo. El gel se comporta como un filtro poroso que retrasa la migración de los fragmentos más grandes. Las moléculas de menor tamaño se mueven a través del gel con mayor rapidez, lo que permite la separación de una mezcla de ácidos nucleicos en función de su tamaño.
En las carreras que se llevan a cabo en los principales hipódromos argentinos se realizan oficialmente controles antidoping de los caballos que obtienen los puestos primero y segundo de cada prueba. Muestras de la orina de esos animales son enviadas a laboratorios donde se realiza el análisis. Los propietarios tienen la posibilidad de apelar si tienen dudas sobre el origen de una muestra de orina que arrojó resultados positivos. Pero mediante los métodos explicados de biología molecular es posible obtener ADN de muestras de sangre y compararlo con el ADN de muestras de orina del animal en cuestión, y así garantizar la correcta identificación de los casos de doping positivo.
Como se aprecia, la cantidad y variedad de información almacenada en la molécula de ADN puede resultar de utilidad para diferentes propósitos. En el ADN existen regiones altamente variables que permiten la identificación individual y regiones menos variables que posibilitan la diferenciación de las especies. Además, la molécula de ADN posee suficiente estabilidad como para permitir el análisis de muestras biológicas con diferente grado de conservación y procesamiento. Estas técnicas moleculares contribuyen a mejorar la calidad de vida y la sanidad humana y de animales.
Lecturas sugeridas
BEARDSLEY S, 2005, 'Bending to bar code', Scientific American, 292, 5: 12-13.
FRANKHAM R, BALLOU JD y BRISCOE DA, 2002, Introduction to conservation genetics, Cambridge University Press.
MAFRA I, FERREIRA IM, FARIA MA y OLIVEIRA B, 2004, 'A novel approach to the quantification of bovine milk in ovine cheeses using a duplex polymerase chain reaction method', Journal of Agricultural and Food Chemistry, 52: 4943-4947.
|
|




