El Comité de Redacción de Acta Bioquímica Clínica Latinoamericana ha seleccionado este artículo de Investigación y Ciencia, julio de 2001, para su difusión en el FABA-Informa
Los responsables de la administración sanitaria y los médicos contemplan con temor fundado la creciente ineficacia de la farmacopea antibiótica. Uno tras otro, el doble centenar de antibióticos va quedando fuera de servicio, por inútil. Las bacterias que sobreviven se hacen más fuertes. Y se propagan. Cada vez hay más cepas resistentes a los antibióticos. La tuberculosis, la meningitis o la neumonía, infecciones que se combatían con antibiótico, no se curan con la facilidad de antaño. Aumenta el número de infecciones bacterianas de pronóstico quizá letal.
Las bacterias son agresores astutos. Además se ha dado y se sigue concediendo, lo que necesitan para su éxito asombroso. Con el uso inadecuado o abusivo de los antibióticos se ha fomentado la evolución de cepas superiores de bacterias. Así ocurre cuando no se completa una tanda de antibióticos, se usan para una infección vírica o se aplican a un mal inadecuado. Se calcula que entre un tercio y la mitad de los antibióticos recetados no son necesarios. Con todo ello, lo único que se consigue es que la bacteria débil muera y la fuente se torne más vigorosa.
Al margen de ese mal uso de la sociedad y de su abuso en clínica, el destino inevitable de los antibióticos es su rápido envejecimiento. Las bacterias -que se multiplican a través de muchas divisiones celulares a lo largo del día- siempre aprenden algo nuevo; algunas de las más fuertes sobrevivirán y prosperarán.
En los últimos diez años, se ha salido por fin de la situación de complacencia en que nos hallábamos sobre el dominio de las infecciones. Laboratorios de titularidad pública y empresas farmacéuticas se han volcado a la investigación antibacteriana. Se ensayan todos los procedimientos imaginables para atacar a las bacterias y se multiplican los antibióticos que se preparan con la información obtenida en el estudio del genoma y de las proteínas.
Pero ni la investigación apasionante ni el desarrollo de fármacos constituyen ninguna panacea. Ahora bien, si se combinan con un uso razonable de los antibióticos pueden prestarnos ayuda. La oficina norteamericana sobre control de alimentación y fármacos (FDA) aprobó en abril del año 2000 el primer tipo nuevo de antibiótico clínico en 35 años; el linezolid. En lista de espera, o en fases previas, hay varios agentes más.
El desmantelamiento de la pared bacteriana
Casi todos los antibióticos que se han desarrollado hasta la fecha proceden de la naturaleza. Los científicos los han identificado y los han refinado, pero no los han creado. Desde el comienzo de la vida en este planeta, los organismos han luchado por los limitados recursos que tenían a su disposición. De esa pugna surgió la evolución de los antibióticos. La capacidad de producir tales compuestos poderosos confiere a un organismo -hongo, planta u otra especie bacteriana- una ventaja sobre las restantes bacterianas sensibles al antibiótico. En esa presión de selección se esconde el motor natural del desarrollo de los antibióticos.
Entre los que han conocido mayor éxito se cuenta la vancomicina, identificada por los laboratorios Eli Lilly en 1956. El comprender su mecanismo de operación ha permitido adentrarnos en el mecanismo de acción de los antibióticos glicopéptidos, una de las siete clases principales. Se trata de un avance importante, por cuanto la vancomicina se ha convertido en el último recurso, el único fármaco eficaz que queda frente a la infección más letal que puede contraerse en el hospital: la del Staphylococcus aureus, resistente a la meticilina. Pero el poder de la vancomicina se encuentra en peligro.
La vancomicina ataca la pared bacteriana que envuelve a la célula y su membrana, confiriéndole estructura y sostén. Ni la vancomicina ni otros fármacos afines dañan las células de humanos y mamíferos, que carecen de tal pared (poseen en cambio un citoesqueleto, una estructura interna que les da consistencia). La pared bacteriana consta fundamentalmente de peptidoglicanos, un material que, de acuerdo con su nombre, contiene péptidos y azúcares. A medida que la célula organiza este material -un proceso constante, porque todo peptidoglicano necesita reemplazarse cuando se degrada-, las unidades glucídicas se unen entre sí mediante la acción de la enzima transglucosidasa y forman una suerte de malla. En esta estructura, una de cada dos unidades de azúcar se une a una cadena peptídica corta. Cada cadena peptídica posee cinco aminoácidos, de los cuales los últimos son una L-lisina y dos D-alaninas. Se encarga la enzima transpeptidasa de reunir las cadenas peptídicas, eliminando a la D-alanina final y uniendo la D-alanina penúltima a una L-lisina de una cadena de azúcares diferente. En razón de ello, las cadenas de glúcidos quedan unidas mediante cadenas peptídicas. Todos estos enlaces cruzados tejen un material muy trenzado, esencial para la supervivencia de la célula; sin él, la célula estallaría por su propia presión interna.
La vancomicina se interpone en la formación de ese material decisivo. El antibiótico se halla preparado para unirse en cadenas peptídicas, antes de que éstas lo hagan entre sí por intervención de la transpeptidasa. El fármaco, al ubicarse de las D-alaninas terminales, evita que la enzima lleve a cabo su tarea. Sin las conexiones entrelazadas, el peptidoglicano se degrada, y la célula estalla y se muere.
El filo de la navaja
Nos enseña la historia de la vancomicina que bastan alteraciones moleculares muy pequeñas para engendrar profundas diferencias. Y las bacterias encuentran múltiples estrategias para engañar a los fármacos, lo que obliga a la búsqueda de medicamentos, nuevos o regenerados. Tradicionalmente, el proceso de identificación de candidatos consistía en un muestreo con células íntegras: las moléculas de interés se aplicaban a células bacterianas vivas. Se trata de un método de probada utilidad, corroborado con el descubrimiento de muchos fármacos, la vancomicina incluida.
Los ensayos que se realizan en dominios moleculares ofrecen una alternativa poderosa. A través de ese muestreo se identifican sólo los compuestos que tienen un mecanismo de acción especificado. Un ejemplo sería la búsqueda específica de inhibidores de la transpeptidasa. Pese a la dificultad que entraña el diseño de tales ensayos, descubre fármacos potenciales con modos de acción conocidos. El problema es que sólo se investiga una enzima cada vez. Se daría un gran paso si se pudiera perseguir simultáneamente más de un objetivo (como ocurre en el proceso en que participan células íntegras) sin merma del conocimiento implícito del mecanismo de operación del fármaco. El gran paso se ha dado. Se ha reconstruido en el tubo de ensayo la vía multienzimática de una bacteria. Con este sistema se pueden identificar moléculas que degradan profundamente una de las enzimas o alteran de un modo sutil varias de ellas.
Con la automatización y la miniaturización se ha multiplicado la celeridad en el cribado de compuestos. La robótica permite estudiar los compuestos a millares. Al mismo tiempo, la miniaturización ha reducido el costo del proceso utilizando cantidades mínimas de reactivos. Con sistemas de separación ultrarrápidos, pueden investigarse cientos de miles de compuestos en un día. Merced a los nuevos métodos de la química combinatoria se pueden diseñar cantidades inmensas de compuestos. En el futuro, algunas de estas moléculas nuevas procederán de las mismas moléculas. Una vez que sea comprendida la vía por la que estos organismos producen antibióticos, la ingeniería genética facilitará la síntesis de nuevas moléculas relacionadas con ellos.
La ventaja de la genómica
El diseño de fármacos y su muestreo se ha beneficiado enormemente del desarrollo reciente de la genómica. Con el conocimiento de los genes y de la síntesis de las proteínas por ellos cifradas, la ciencia se ha adentrado en las propias entrañas moleculares del organismo. A la manera de un servicio de contraespionaje microbiano, importa ahora atentar contra genes de importancia capital, bloquear la síntesis de una proteína específica o alterar la capacidad de un organismo para infectar o desarrollar resistencia.
Muchos de los objetivos contra los que van dirigidos los antibióticos son genes esenciales, genes que provocan la muerte celular si dejan de funcionar.
Las proteínas codificadas por los genes esenciales no son los únicos objetivos moleculares en la búsqueda de antibióticos. Importan también los genes que codifican factores de virulencia; eluden éstos la respuesta inmunitaria del huésped y de ese modo preparan el terreno para la colonización de la bacteria. Antes resultaba muy difícil identificar tales genes porque se "activan" o transcriben por sucesos en el tejido del huésped cuya reproducción in vitro se hacía muy compleja. Mas con la técnica actual de expresión in vivo (IVET) se puede insertar una secuencia singular de ADN, una forma de etiqueta que desactiva un gen, en cada gen bacteriano. Se aplican bacterias etiquetadas para infectar un organismo, se recuperan luego y se identifican las etiquetas. La desaparición de cualquier etiqueta significa que los genes a los que estaban unidas eran esenciales para la supervivencia de la bacteria, tan esenciales que la bacteria no podría sin ellos vivir en el huésped.
Desde hace tiempo se esperaba que la identificación e inhibición de los factores de virulencia permitieran al sistema inmunitario del organismo combatir las bacterias patogénicas antes de su instalación. Algo se ha avanzado. En un estudio reciente, una molécula experimental que inhibe el factor de virulencia de S. Aureus ha servido para que los ratones de prueba resistieran la infección.
Además de investigar los genes esenciales y los factores de virulencia, los laboratorios empiezan a descubrir qué genes confieren resistencia a los antibióticos. La polarización en ellos del ataque ayudará a rejuvenecer antibióticos desechados por ineficaces. En esta línea ejercen su acción los antibióticos b-lactámicos como la penicilina. El mecanismo habitual de resistencia contra los antibióticos b-lactámicos radica en la producción bacteriana de b-lactamasa, enzima que corta uno de los enlaces del antibiótico, cambiando su estructura y evitando la inhibición de la transpeptidasa. Si se silencia la b-lactamasa, el antibiótico permanece eficaz. Eso es justamente lo que hace el ácido clavulánico, un inhibidor de la b-lactamasa: se mezcla con la amoxicilina para crear el antibiótico Augmentine.
Cuando en un futuro se domine mejor la trascripción del ADN, será práctica rutinaria la identificación de determinantes de resistencia, como la b-lactamasa, y factores de virulencia. Habrá llegado entonces el momento de poder identificar los genes que intervienen en diferentes condiciones de desarrollo celular. Mediante la identificación de los genes bacterianos cuya expresión aumenta al infectar un huésped se determinarán los genes de virulencia. Se establecerán los genes de resistencia a los antibióticos mediante la comparación entre niveles de expresión en bacterias tratadas con antibióticos y los manifestados en bacterias sin tratar. Esta técnica ya ha detectado cambios minúsculos en el número de episodios de transcripción. Con el dominio del perfil de transcripción del ADN, podrá establecerse si determinados fármacos aplican mecanismos de acción totalmente nuevos o tienen blancos celulares inéditos que podrían señalar caminos a la investigación antibiótica no transitados.
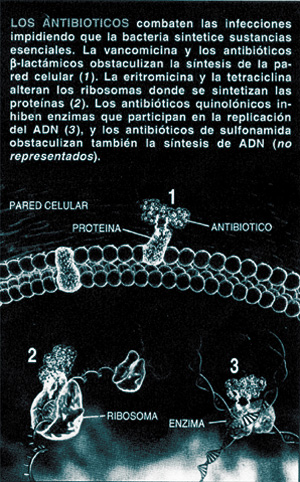
Sacrificio del mensajero
En otra línea interesante de la investigación genómica se busca la inoperancia del ARN bacteriano. En su mayor proporción, el ARN es ribosómico (ARNr), componente estructural principal de los ribosomas. Son éstos fábricas de ensamblaje de las proteínas. La vulnerabilidad del ARN ribosómico yace en la diversidad de lugares donde pueden anclarse los fármacos. Carece, además, de capacidad para autorrepararse. En 1987 se comprobó que los antibióticos aminoglicósidos -la estreptomicina, entre ellos- se unían al ARNr, lo que determinaba que el ribosoma errase en la lectura del código correspondiente al ensamblaje de la proteína. Muchos de estos antibióticos, sin embargo, además de ser tóxicos, presentan una utilidad limitada. En el Instituto Scripps de Investigaciones de La Jolla se acaba de observar un nuevo dímero de aminoglicósido sintético que muestra menor toxicidad.
Se puede entorpecer la acción del ARN mensajero (ARNm) que dirige la síntesis de proteínas y cursa entre el código genético y el ribosoma. El ARN mensajero se crea mediante la lectura de una de las hebras del ADN, aplicando las mismas interacciones entre ácidos nucleicos, o pares de bases, que mantiene unida la doble hélice. La molécula de ARNm lleva entonces el mensaje al ribosoma, donde se sintetiza una proteína a través del proceso de traducción. Puesto que cada ARNm codifica una proteína específica y difiere de otros ARNm, se cuenta con la posibilidad de forjar otras interacciones entre pequeñas moléculas orgánicas -esto es, no proteínas- y ARNm específicos. Es ni más ni menos lo que se les ha ocurrido a químicos de Parke-Davis para combatir la infección de VIH. Tras identificar moléculas que se unen a una parte de una secuencia de ARNm, evitan que ésta interaccione con una proteína activadora necesaria, inhibiendo así la replicación del VIH. Semejante logro experimental debería incitar nuevos estudios sobre el ARNm en la búsqueda de fármacos.
Se trabaja con similar empeño en el dominio de la terapia antisentido. Al generar secuencias de nucleótidos con una secuencia específica de ARNm, encierran dicho ácido nucleico. De esta manera, el ARNm no puede liberarse del fármaco; o se destruye o queda inactivo. Aunque la FDA ha aprobado ya el primer fármaco antisentido para infecciones del citomegalovirus en el hombre, este tipo de medicamentos no han tenido éxito en las infecciones bacterianas, por diversas razones, entre ellas la toxicidad y la dificultad de que el fármaco alcance una concentración adecuada en el sitio de la infección. Pese a todo, se trata de un campo promisorio.
De lo que no cabe duda es de que todos estos planteamientos genómicos facilitan la identificación y evaluación de una gama de objetivos biológicos contra los que pueden dirigirse moléculas eficaces. Quedan descartados, por nocivos, numerosos antibióticos desarrollados en el siglo XX. Pero al comparar la secuencia génica del objetivo potencial con los genes que se encuentran en el hombre, se pueden identificar genes exclusivos de las bacterias; una vez acotados, se puede concentrar la atención sobre ellos. Igualmente, al comparar la secuencia génica de un objetivo con las de otras bacterias, es posible evaluar la selectividad de un fármaco. Una secuencia objetivo que aparece en todas las bacterias habría de generar, con probabilidad, un antibiótico activo contra muchas bacterias diferentes; vale decir, un antibiótico de amplio espectro. Por el contrario, una secuencia objetivo que aparece sólo en el genoma de algunas bacterias generaría un antibiótico de margen estrecho.
Si los médicos descubren en una fase temprana la cepa bacteriana causante de la infección, pueden afinar y recetar un antibiótico de espectro limitado. Puesto que el medicamento afectará sólo a un subgrupo de la población bacteriana, la presión de selección para desarrollar resistencia será escasa. Los avances en replicación rápida del ADN y en la obtención de perfiles de transcripción podrán hacer que pronto la identificación de cepas bacterianas constituya un protocolo rutinario. Aunque el cuadro parezca ahora más nítido que años atrás, conviene no perder de vista que, en biología, la carrera armamentística no es cosa nueva. A cada contraataque del hombre le seguirá una respuesta de la bacteria, del tipo que sea; a veces le basta con cambiar un átomo de un aminoácido.
Los responsables de la administración sanitaria y los médicos contemplan con temor fundado la creciente ineficacia de la farmacopea antibiótica. Uno tras otro, el doble centenar de antibióticos va quedando fuera de servicio, por inútil. Las bacterias que sobreviven se hacen más fuertes. Y se propagan. Cada vez hay más cepas resistentes a los antibióticos. La tuberculosis, la meningitis o la neumonía, infecciones que se combatían con antibiótico, no se curan con la facilidad de antaño. Aumenta el número de infecciones bacterianas de pronóstico quizá letal.
Las bacterias son agresores astutos. Además se ha dado y se sigue concediendo, lo que necesitan para su éxito asombroso. Con el uso inadecuado o abusivo de los antibióticos se ha fomentado la evolución de cepas superiores de bacterias. Así ocurre cuando no se completa una tanda de antibióticos, se usan para una infección vírica o se aplican a un mal inadecuado. Se calcula que entre un tercio y la mitad de los antibióticos recetados no son necesarios. Con todo ello, lo único que se consigue es que la bacteria débil muera y la fuente se torne más vigorosa.
Al margen de ese mal uso de la sociedad y de su abuso en clínica, el destino inevitable de los antibióticos es su rápido envejecimiento. Las bacterias -que se multiplican a través de muchas divisiones celulares a lo largo del día- siempre aprenden algo nuevo; algunas de las más fuertes sobrevivirán y prosperarán.
En los últimos diez años, se ha salido por fin de la situación de complacencia en que nos hallábamos sobre el dominio de las infecciones. Laboratorios de titularidad pública y empresas farmacéuticas se han volcado a la investigación antibacteriana. Se ensayan todos los procedimientos imaginables para atacar a las bacterias y se multiplican los antibióticos que se preparan con la información obtenida en el estudio del genoma y de las proteínas.
Pero ni la investigación apasionante ni el desarrollo de fármacos constituyen ninguna panacea. Ahora bien, si se combinan con un uso razonable de los antibióticos pueden prestarnos ayuda. La oficina norteamericana sobre control de alimentación y fármacos (FDA) aprobó en abril del año 2000 el primer tipo nuevo de antibiótico clínico en 35 años; el linezolid. En lista de espera, o en fases previas, hay varios agentes más.
El desmantelamiento de la pared bacteriana
Casi todos los antibióticos que se han desarrollado hasta la fecha proceden de la naturaleza. Los científicos los han identificado y los han refinado, pero no los han creado. Desde el comienzo de la vida en este planeta, los organismos han luchado por los limitados recursos que tenían a su disposición. De esa pugna surgió la evolución de los antibióticos. La capacidad de producir tales compuestos poderosos confiere a un organismo -hongo, planta u otra especie bacteriana- una ventaja sobre las restantes bacterianas sensibles al antibiótico. En esa presión de selección se esconde el motor natural del desarrollo de los antibióticos.
Entre los que han conocido mayor éxito se cuenta la vancomicina, identificada por los laboratorios Eli Lilly en 1956. El comprender su mecanismo de operación ha permitido adentrarnos en el mecanismo de acción de los antibióticos glicopéptidos, una de las siete clases principales. Se trata de un avance importante, por cuanto la vancomicina se ha convertido en el último recurso, el único fármaco eficaz que queda frente a la infección más letal que puede contraerse en el hospital: la del Staphylococcus aureus, resistente a la meticilina. Pero el poder de la vancomicina se encuentra en peligro.
La vancomicina ataca la pared bacteriana que envuelve a la célula y su membrana, confiriéndole estructura y sostén. Ni la vancomicina ni otros fármacos afines dañan las células de humanos y mamíferos, que carecen de tal pared (poseen en cambio un citoesqueleto, una estructura interna que les da consistencia). La pared bacteriana consta fundamentalmente de peptidoglicanos, un material que, de acuerdo con su nombre, contiene péptidos y azúcares. A medida que la célula organiza este material -un proceso constante, porque todo peptidoglicano necesita reemplazarse cuando se degrada-, las unidades glucídicas se unen entre sí mediante la acción de la enzima transglucosidasa y forman una suerte de malla. En esta estructura, una de cada dos unidades de azúcar se une a una cadena peptídica corta. Cada cadena peptídica posee cinco aminoácidos, de los cuales los últimos son una L-lisina y dos D-alaninas. Se encarga la enzima transpeptidasa de reunir las cadenas peptídicas, eliminando a la D-alanina final y uniendo la D-alanina penúltima a una L-lisina de una cadena de azúcares diferente. En razón de ello, las cadenas de glúcidos quedan unidas mediante cadenas peptídicas. Todos estos enlaces cruzados tejen un material muy trenzado, esencial para la supervivencia de la célula; sin él, la célula estallaría por su propia presión interna.
La vancomicina se interpone en la formación de ese material decisivo. El antibiótico se halla preparado para unirse en cadenas peptídicas, antes de que éstas lo hagan entre sí por intervención de la transpeptidasa. El fármaco, al ubicarse de las D-alaninas terminales, evita que la enzima lleve a cabo su tarea. Sin las conexiones entrelazadas, el peptidoglicano se degrada, y la célula estalla y se muere.
El filo de la navaja
Nos enseña la historia de la vancomicina que bastan alteraciones moleculares muy pequeñas para engendrar profundas diferencias. Y las bacterias encuentran múltiples estrategias para engañar a los fármacos, lo que obliga a la búsqueda de medicamentos, nuevos o regenerados. Tradicionalmente, el proceso de identificación de candidatos consistía en un muestreo con células íntegras: las moléculas de interés se aplicaban a células bacterianas vivas. Se trata de un método de probada utilidad, corroborado con el descubrimiento de muchos fármacos, la vancomicina incluida.
Los ensayos que se realizan en dominios moleculares ofrecen una alternativa poderosa. A través de ese muestreo se identifican sólo los compuestos que tienen un mecanismo de acción especificado. Un ejemplo sería la búsqueda específica de inhibidores de la transpeptidasa. Pese a la dificultad que entraña el diseño de tales ensayos, descubre fármacos potenciales con modos de acción conocidos. El problema es que sólo se investiga una enzima cada vez. Se daría un gran paso si se pudiera perseguir simultáneamente más de un objetivo (como ocurre en el proceso en que participan células íntegras) sin merma del conocimiento implícito del mecanismo de operación del fármaco. El gran paso se ha dado. Se ha reconstruido en el tubo de ensayo la vía multienzimática de una bacteria. Con este sistema se pueden identificar moléculas que degradan profundamente una de las enzimas o alteran de un modo sutil varias de ellas.
Con la automatización y la miniaturización se ha multiplicado la celeridad en el cribado de compuestos. La robótica permite estudiar los compuestos a millares. Al mismo tiempo, la miniaturización ha reducido el costo del proceso utilizando cantidades mínimas de reactivos. Con sistemas de separación ultrarrápidos, pueden investigarse cientos de miles de compuestos en un día. Merced a los nuevos métodos de la química combinatoria se pueden diseñar cantidades inmensas de compuestos. En el futuro, algunas de estas moléculas nuevas procederán de las mismas moléculas. Una vez que sea comprendida la vía por la que estos organismos producen antibióticos, la ingeniería genética facilitará la síntesis de nuevas moléculas relacionadas con ellos.
La ventaja de la genómica
El diseño de fármacos y su muestreo se ha beneficiado enormemente del desarrollo reciente de la genómica. Con el conocimiento de los genes y de la síntesis de las proteínas por ellos cifradas, la ciencia se ha adentrado en las propias entrañas moleculares del organismo. A la manera de un servicio de contraespionaje microbiano, importa ahora atentar contra genes de importancia capital, bloquear la síntesis de una proteína específica o alterar la capacidad de un organismo para infectar o desarrollar resistencia.
Muchos de los objetivos contra los que van dirigidos los antibióticos son genes esenciales, genes que provocan la muerte celular si dejan de funcionar.
Las proteínas codificadas por los genes esenciales no son los únicos objetivos moleculares en la búsqueda de antibióticos. Importan también los genes que codifican factores de virulencia; eluden éstos la respuesta inmunitaria del huésped y de ese modo preparan el terreno para la colonización de la bacteria. Antes resultaba muy difícil identificar tales genes porque se "activan" o transcriben por sucesos en el tejido del huésped cuya reproducción in vitro se hacía muy compleja. Mas con la técnica actual de expresión in vivo (IVET) se puede insertar una secuencia singular de ADN, una forma de etiqueta que desactiva un gen, en cada gen bacteriano. Se aplican bacterias etiquetadas para infectar un organismo, se recuperan luego y se identifican las etiquetas. La desaparición de cualquier etiqueta significa que los genes a los que estaban unidas eran esenciales para la supervivencia de la bacteria, tan esenciales que la bacteria no podría sin ellos vivir en el huésped.
Desde hace tiempo se esperaba que la identificación e inhibición de los factores de virulencia permitieran al sistema inmunitario del organismo combatir las bacterias patogénicas antes de su instalación. Algo se ha avanzado. En un estudio reciente, una molécula experimental que inhibe el factor de virulencia de S. Aureus ha servido para que los ratones de prueba resistieran la infección.
Además de investigar los genes esenciales y los factores de virulencia, los laboratorios empiezan a descubrir qué genes confieren resistencia a los antibióticos. La polarización en ellos del ataque ayudará a rejuvenecer antibióticos desechados por ineficaces. En esta línea ejercen su acción los antibióticos b-lactámicos como la penicilina. El mecanismo habitual de resistencia contra los antibióticos b-lactámicos radica en la producción bacteriana de b-lactamasa, enzima que corta uno de los enlaces del antibiótico, cambiando su estructura y evitando la inhibición de la transpeptidasa. Si se silencia la b-lactamasa, el antibiótico permanece eficaz. Eso es justamente lo que hace el ácido clavulánico, un inhibidor de la b-lactamasa: se mezcla con la amoxicilina para crear el antibiótico Augmentine.
Cuando en un futuro se domine mejor la trascripción del ADN, será práctica rutinaria la identificación de determinantes de resistencia, como la b-lactamasa, y factores de virulencia. Habrá llegado entonces el momento de poder identificar los genes que intervienen en diferentes condiciones de desarrollo celular. Mediante la identificación de los genes bacterianos cuya expresión aumenta al infectar un huésped se determinarán los genes de virulencia. Se establecerán los genes de resistencia a los antibióticos mediante la comparación entre niveles de expresión en bacterias tratadas con antibióticos y los manifestados en bacterias sin tratar. Esta técnica ya ha detectado cambios minúsculos en el número de episodios de transcripción. Con el dominio del perfil de transcripción del ADN, podrá establecerse si determinados fármacos aplican mecanismos de acción totalmente nuevos o tienen blancos celulares inéditos que podrían señalar caminos a la investigación antibiótica no transitados.

Sacrificio del mensajero
En otra línea interesante de la investigación genómica se busca la inoperancia del ARN bacteriano. En su mayor proporción, el ARN es ribosómico (ARNr), componente estructural principal de los ribosomas. Son éstos fábricas de ensamblaje de las proteínas. La vulnerabilidad del ARN ribosómico yace en la diversidad de lugares donde pueden anclarse los fármacos. Carece, además, de capacidad para autorrepararse. En 1987 se comprobó que los antibióticos aminoglicósidos -la estreptomicina, entre ellos- se unían al ARNr, lo que determinaba que el ribosoma errase en la lectura del código correspondiente al ensamblaje de la proteína. Muchos de estos antibióticos, sin embargo, además de ser tóxicos, presentan una utilidad limitada. En el Instituto Scripps de Investigaciones de La Jolla se acaba de observar un nuevo dímero de aminoglicósido sintético que muestra menor toxicidad.
Se puede entorpecer la acción del ARN mensajero (ARNm) que dirige la síntesis de proteínas y cursa entre el código genético y el ribosoma. El ARN mensajero se crea mediante la lectura de una de las hebras del ADN, aplicando las mismas interacciones entre ácidos nucleicos, o pares de bases, que mantiene unida la doble hélice. La molécula de ARNm lleva entonces el mensaje al ribosoma, donde se sintetiza una proteína a través del proceso de traducción. Puesto que cada ARNm codifica una proteína específica y difiere de otros ARNm, se cuenta con la posibilidad de forjar otras interacciones entre pequeñas moléculas orgánicas -esto es, no proteínas- y ARNm específicos. Es ni más ni menos lo que se les ha ocurrido a químicos de Parke-Davis para combatir la infección de VIH. Tras identificar moléculas que se unen a una parte de una secuencia de ARNm, evitan que ésta interaccione con una proteína activadora necesaria, inhibiendo así la replicación del VIH. Semejante logro experimental debería incitar nuevos estudios sobre el ARNm en la búsqueda de fármacos.
Se trabaja con similar empeño en el dominio de la terapia antisentido. Al generar secuencias de nucleótidos con una secuencia específica de ARNm, encierran dicho ácido nucleico. De esta manera, el ARNm no puede liberarse del fármaco; o se destruye o queda inactivo. Aunque la FDA ha aprobado ya el primer fármaco antisentido para infecciones del citomegalovirus en el hombre, este tipo de medicamentos no han tenido éxito en las infecciones bacterianas, por diversas razones, entre ellas la toxicidad y la dificultad de que el fármaco alcance una concentración adecuada en el sitio de la infección. Pese a todo, se trata de un campo promisorio.
De lo que no cabe duda es de que todos estos planteamientos genómicos facilitan la identificación y evaluación de una gama de objetivos biológicos contra los que pueden dirigirse moléculas eficaces. Quedan descartados, por nocivos, numerosos antibióticos desarrollados en el siglo XX. Pero al comparar la secuencia génica del objetivo potencial con los genes que se encuentran en el hombre, se pueden identificar genes exclusivos de las bacterias; una vez acotados, se puede concentrar la atención sobre ellos. Igualmente, al comparar la secuencia génica de un objetivo con las de otras bacterias, es posible evaluar la selectividad de un fármaco. Una secuencia objetivo que aparece en todas las bacterias habría de generar, con probabilidad, un antibiótico activo contra muchas bacterias diferentes; vale decir, un antibiótico de amplio espectro. Por el contrario, una secuencia objetivo que aparece sólo en el genoma de algunas bacterias generaría un antibiótico de margen estrecho.
Si los médicos descubren en una fase temprana la cepa bacteriana causante de la infección, pueden afinar y recetar un antibiótico de espectro limitado. Puesto que el medicamento afectará sólo a un subgrupo de la población bacteriana, la presión de selección para desarrollar resistencia será escasa. Los avances en replicación rápida del ADN y en la obtención de perfiles de transcripción podrán hacer que pronto la identificación de cepas bacterianas constituya un protocolo rutinario. Aunque el cuadro parezca ahora más nítido que años atrás, conviene no perder de vista que, en biología, la carrera armamentística no es cosa nueva. A cada contraataque del hombre le seguirá una respuesta de la bacteria, del tipo que sea; a veces le basta con cambiar un átomo de un aminoácido.